Jean-François Vibert : biographie d’un passeur
Si les progrès de la médecine ne sont plus à démontrer, moins connus du public sont en revanche les apports des mathématiques et de la modélisation à ces progrès. Des pionniers comme le professeur Jean-François Vibert, à la fois médecin (neurophysiologiste) et professeur de bio-mathématique au sein du labo, ont largement contribué par leur curiosité intellectuelle et leur esprit d’innovation à cette alliance. Aujourd’hui Professeur émérite des universités à l’
hôpital Saint-Antoine, Jean-François Vibert a réalisé l’essentiel de ses recherches dans le cadre de de l’unité de recherche Inserm U707 (épidémiologie, sciences de l’information et modélisation). Il s’est consacré́ à la compréhension de la neurogenèse du rythme respiratoire à l’aide de réseaux de neurones, puis à la modélisation de la diffusion des informations dans les systèmes dynamiques complexes. Développeur d’un logiciel de simulation de réseau de neurones (XNBC), il a contribué en 1984 au réseau « sentinelle » de modélisation des épidémies (grippe, hépatites, rougeole…) fondé par Alain-Jacques Valleron, aujourd’hui reconnu d’utilité publique dans un contexte de regain épidémique. Son fils, Eric Vibert, professeur de chirurgie hépato-biliaire à l’hôpital Paul Brousse, en créant en 2020 la chaire d’innovation Bopa (bloc opératoire augmenté) se situe dans la lignée de cet esprit d’exploration qui contribue à repousser sans cesse les frontières de la connaissance. C’est cette histoire que nous proposons brièvement de retracer ici.
Déplacer les frontières, faire dialoguer les disciplines
Dans les années 1970, Jean-François Vibert travaille en tant que jeune assistant dans un laboratoire de neurophysiologie de l’hôpital Saint Antoine où on s’intéresse à la genèse du rythme respiratoire au niveau du tronc cérébral. C’est donc dans ce contexte qu’un rapprochement va s’opérer entre neurophysiologie et réseaux de neurones. Dans ce laboratoire on procédait à des manipulations sur les chats : des électrodes étaient fichées au niveau du cerveau (au niveau du tronc cérébral) pour enregistrer les cellules (neurones) qui sont en rapport avec la respiration. Cela permettait d’enregistrer le nerf phrénique (qui commande le diaphragme), l’inspiration, l’expiration, autrement dit d’obtenir le timing de la respiration. Or, ce laboratoire était un des tout premiers qui était parvenu à acheter un ordinateur, grâce auquel on pouvait traiter en temps réel les signaux enregistrés depuis les électrodes. La décharge des neurones ainsi capturée était corrélée avec l’enregistrement du nerf phrénique. Cela permis notamment de découvrir qu’il y avait des cellules répondant à l’inspiration, d’autres à l’expiration, puis d’autres qui étaient ce qu’on appelait IE (inspiratoires-expiratoires). Celles-ci commençaient à décharger au milieu de l’inspiration et s’arrêtaient doucement vers le milieu de l’expiration, se trouvant ainsi à cheval sur les deux temps de la respiration. « A l’époque, on ne savait pas très bien où se trouvaient vraiment les centres respiratoires, mais on en avait quand même une idée car certains physiologistes pensaient que c’était au niveau du tronc cérébral, c’est la raison pour laquelle on allait piquer nos électrodes à cet endroit. A cette occasion, l’équipe avait d’ailleurs fait un atlas et une reconstruction 3D des centres respiratoires, qui avait d’ailleurs fait l’objet d’un film au CNRS (c’était un labo CNRS à l’époque). On essayait de comprendre comment la respiration pouvait être un mouvement lent (on respire 10 à 12 fois / min) alors que les cellules et les neurones déchargeaient à toute vitesse. » Jean-François Vibert, s’intéressant depuis toujours à l’électronique, « à tout ce qui était un peu technique », se mit à alors à la programmation sur l’ordinateur du laboratoire et eut l’idée de développer un logiciel de simulation de réseaux de neurones pour comprendre ce phénomène. C’est dans ce cadre qu’il développa le logiciel, l’outil de neurobiologie numérique XNBC, qui permet de modéliser les neurones, de plusieurs façons, de les assembler en réseaux, afin de voir ce qui se passe.
De la tête d’un chat à l’embryon du poulet : le réseau de neurones XNBC
Ce logiciel permet de simuler les neurones de deux façons : soit relativement simple, soit plus complexe avec, en entrées, tous les courants (sodium, potassium, etc.) qui traversent la membrane, et qui génèrent les potentiels d’action. Le réseau de neurones XNBC est constitué d’un ou plusieurs noyaux et/ou d’un ou plusieurs neurones isolés. Les noyaux (groupe de neurones, chacun appartenant à un cluster donné) et les neurones peuvent être positionnés anatomiquement si nécessaire. « Les noyaux et les neurones sont reliés entre eux par des liens représentant les axones des neurones constitutifs. Les neurones peuvent être connectés entre eux. Les connexions peuvent être soit excitatrices, soit inhibitrices, soit avec NMDA (excitation de longue durée), soit un mélange d’excitateurs et d’inhibiteurs, appelé connexion aléatoire. La transmission inter neuronale des potentiels d’action, appelée délai interne (ou longueur de l’axone) peut être ajustée, ainsi que le nombre de boutons synaptiques à l’extrémité de l’axone, appelé également poids synaptique. La matrice de connexion peut être définie globalement ou individuellement, neurone par neurone. Les paramètres des neurones et du réseau peuvent être modifiés au cours de la simulation, afin d’imiter les stimulations électriques et l’action des médicaments. »¹.
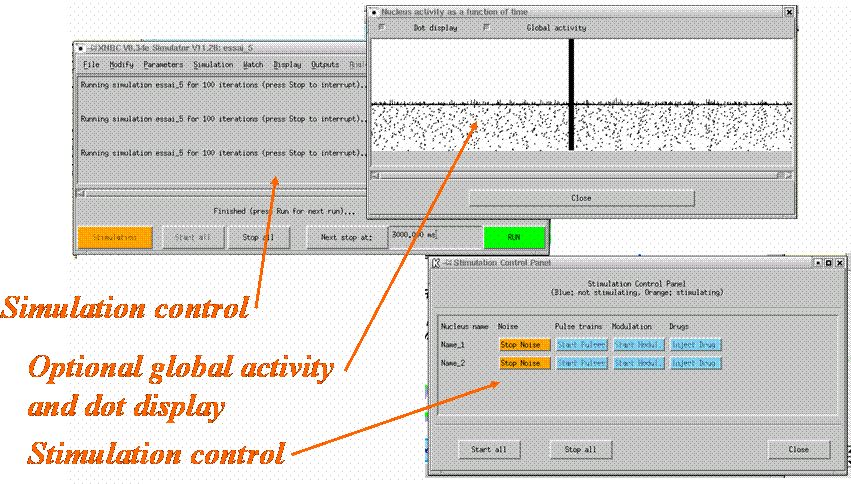
Cet outil permet à la fois de visualiser et d’analyser l’activité du réseau.
Le but de l’usage des réseaux de neurones est donc la simulation, la modélisation permettant de prévoir les effets de telle ou telle manipulation. Les réseaux de neurones permettent ainsi de tester des hypothèses. C’est ce qui arriva, dans les années 1980, lorsque Jean-François Vibert développa avec un de ses étudiants une simulation de la rétine, qui permit de montrer que l’organisation de celle-ci faisait émerger des images. Cette simulation fut ensuite appliquée avec succès à la détection des microcalcifications dans les mammographies. Ou encore, en 2000, avec la « Modélisation du réseau de neurone à l’origine des rythmes chez l’embryon de poulet »².
La sinusoïde bruitée de la queue de l’écrevisse
A partir des années 1990, Jean-François Vibert travaille avec un mathématicien, Khashayar Pakdaman, dont il dirige la thèse. Cette collaboration va permettre d’améliorer les réseaux de neurones. Dans le prolongement de ces travaux, il étudie le codage des neurones avec le Pr J.-P. Segundo au Brain Research Institute de Los Angeles, plus précisément la manière dont les informations sensorielles ou motrices sont codées par les neurones ainsi que le comportement de ces réseaux en présence du bruit. L’objet d’étude était les stretch receptors de l’écrevisse, en français « récepteurs à l’étirement » situés dans la queue de l’écrevisse (récepteur d’une fibre musculaire qu’on a dans tous les muscles qui mesure la longueur du muscle). Il s’agissait de déterminer la manière dont le neurone d’entrée transmettait l’information au neurone de sortie (derrière). L’un des résultats fut de démontrer que le bruit linéarise la réponse. En effet, lorsqu’on regarde la manière dont le stretch receptor répond, on s’aperçoit que si on envoie une sinusoïde « bien propre » la réponse n’est pas du tout linéaire, alors que si on envoie une sinusoïde bruitée, la réponse est linéarisée. Le bruit évite ainsi que les neurones ne se synchronisent simultanément, c’est-à-dire ne se trouvent tous simultanément en phase réfractaire absolue puis relative, empêchant alors le système de répondre³.
What the Frog’s eye tells the frog’s brain?⁴
En conclusion, l’IA (en l’occurrence les réseaux de neurones) s’est donc révélée principalement utile en tant qu’outil de simulation. On est proche de l’usage qu’on peut faire des jumeaux numériques en médecine, un des objets sur lesquels le fils de Jean-François Vibert, Éric Vibert, travaille actuellement au sein de la chaire d’innovation Bopa. Les travaux de ce dernier, ainsi que ceux du Dr Nicolas Golse, sont destinés à tirer profit de tous les outils qu’offre l’IA (via la réalité augmentée par exemple) pour la chirurgie du foie⁵.
Est-ce qu’un jour l’activité neurophysiologique pourra entièrement être décrite et simulée grâce à ces outils ? Jean-François Vibert souligne que les simulateurs sont toujours hyper simplifiés par rapport à la biologie. Même en mettant une dizaine de courants différents, qui ont des constantes de temps différentes, etc., et, même avec un nombre de courants relativement important, ces modèles restent loin de la réalité. En effet, quantité d’éléments ne sont pas pris en compte, en particulier le milieu extérieur des neurones. Si on tient bien compte des conductances de potassium, sodium, calcium, etc, au niveau de la membrane pour générer le potentiel d’action, on néglige en revanche le fait qu’il y a probablement une accumulation de potassium ou de sodium à l’extérieur du neurone qui va probablement moduler les choses (ce que les recherches évoquées plus haut sur la simulation de la rétine ont illustré). Jean-François Vibert pense qu’on n’arrivera jamais exactement à simuler le phénomène biologique, mais sans dogmatisme toutefois, car les progrès fulgurants de l’IA en termes de vitesse de calculs notamment, ne permettent pas selon lui de poser un jugement définitif.
L’inventeur de la théorie des neurones formels, Warren McCulloch, était lui-même, faut-il le rappeler, neurophysiologiste. Son principal collaborateur était le jeune mathématicien Walter Pitts. L’expérience de Jean-François Vibert illustre et actualise une fois encore l’importance de cette collaboration disciplinaire. Elle met notamment en évidence le rôle crucial pour l’IA de se confronter en permanence aux défis de la complexité du vivant. L’aventure continue donc.
¹ https://xnbc.sorbonne-universite.fr.
² « Modélisation du réseau de neurone à l’origine des rythmes chez l’embryon de poulet », Automatique, Biologie et Santé : Modélisation et commande de régulations biologiques, Vol. 9, 2000, p.101-117.
³ J.-F. Vibert et al., “Single Neuron with Recurrent Excitation: Effect of the Transmission Delay”, Neural Networks, 9-5, July 1996, 797-818 et J.-F. Vibert et al., Neurophysiologie: De la physiologie à l’exploration fonctionnelle, Elsevier, 2019.
⁴ McCulloch W.S., Lettvin J.Y, Maturana H.R, Pitts W.H, “What the Frog’s eye tells the frog’s brain”. In : Proc. of the I.R.E. Vol.47 (11), 1959.
⁵ Golse N., Modélisation anatomique, hémodynamique et physiologique en chirurgie hépatique, Université Paris-Saclay, 2021
Bibliographie
Brunet J.-N., Mendizabal A. et al., “Physics-based Deep Neural Network for Augmented Reality during Liver Surgery”, Medical Image Computing and Computer Assisted Intervention – MICCAI 2019. MICCAI 2019. Lecture Notes in Computer Science (), vol 11768. Springer, Cham.
J.-F. Vibert et al., “Single Neuron with Recurrent Excitation: Effect of the Transmission Delay”, Neural Networks, 9-5, July 1996, 797-818.
J.-F. Vibert et al., “XNBC : a simulation tool. Application to the study of neural coding using hybrid netwoks”, BioSystems 40, 1997, 211-218.
J.-F. Vibert et al., “XNBC V9: A user friendly simulation and analysis tool for neurobiologists”, Neurocomputing, 38-40, 2001, 1715-1723.
J.-F. Vibert et al., Neurophysiologie : De la physiologie à l’exploration fonctionnelle, Elsevier, 2019.