Together AI, une startup de recherche en IA basée à San Francisco qui s'est donnée pour mission de faire avancer les frontières de l’IA open source, a récemment publié EVO, un modèle de fondation génomique qui permet des tâches de prédiction et de génération de l'échelle moléculaire à l'échelle du génome, développé en collaboration avec Arc Institute, une organisation de recherche à but non lucratif basée à Palo Alto, près de San Francisco.
Le génome est une séquence qui code complètement l’ADN, l’ARN et les protéines qui orchestrent le fonctionnement d'un organisme. La modélisation des génomes entiers se heurte à deux défis majeurs: les séquences d’ADN sont extrêmement longues (jusqu’à des milliards de nucléotides) et la sensibilité requise pour comprendre pleinement les effets de l’évolution (qui se produit un nucléotide à la fois). En conséquence, les avancées existantes dans la modélisation de séquences biologiques avec l’IA se sont plutôt concentrées sur des capacités spécifiques à une tâche ou à modalité unique.
Les chercheurs de Together AI et de l'Arc Institute ont cherché à surmonter ces difficultés inhérentes à la modélisation de génomes entiers pour développer un modèle qui puisse comprendre non seulement les composants individuels de l’ADN, de l’ARN et des protéines, mais aussi comment ils interagissent pour créer des systèmes complexes.
Plutôt que l'architecture Transformer habituellement utilisée, ils ont opté pour l’architecture hybride StripedHyena, une architecture de traitement du signal en profondeur récemment introduite par Together Research, combinant l'attention rotative et les opérateurs d'hyène pour traiter et rappeler efficacement des modèles dans de longues séquences. Grâce à ses capacités de long contexte et son mode d’inférence efficace, elle leur a permis de générer des séquences allant jusqu’à 650k sur un seul GPU.
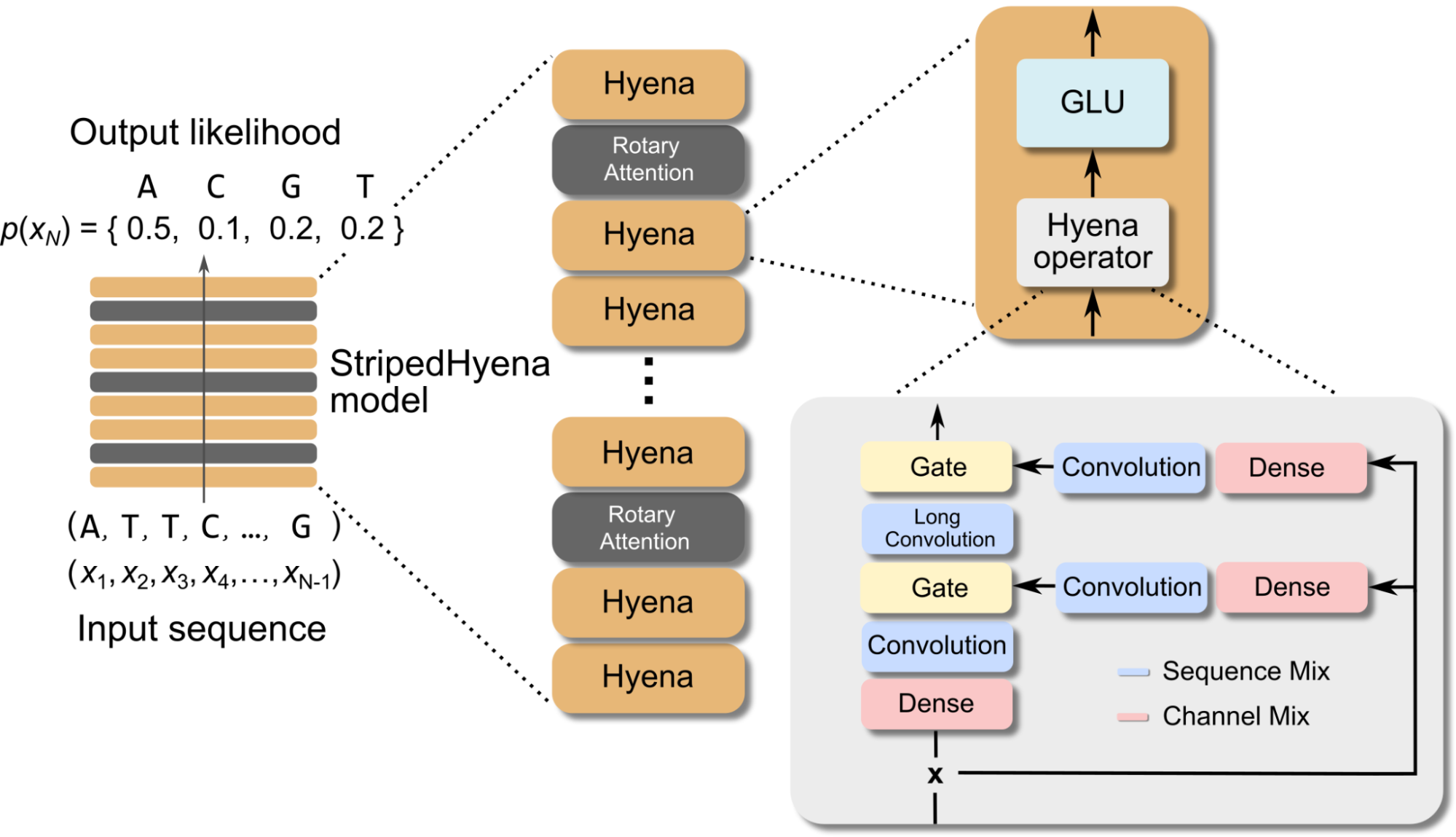
Architecture d'EVO
Les performances du modèle EVO 1
Le modèle EVO 1, qui compte 7 milliards de paramètres, a été entraîné sur OpenGenome, un ensemble de données procaryotes sur le génome entier contenant environ 300 milliards de jetons. Il peut non seulement générer à l’échelle de plusieurs molécules (protéines et ARNnc), mais il a également le potentiel de générer des séquences à l’échelle des génomes entiers :- Prédiction zéro-shot : Evo peut prédire la fonction biologique à l'échelle du génome entier, surpassant même les expériences d'essentialité génétique en laboratoire en un fraction du temps requis ;
- Prédiction multimodale : Evo peut prédire la fonction des protéines, de l'ARN non codant et de l'ADN régulateur, couvrant ainsi toutes les modalités biologiques essentielles ;
- Génération génétique : Evo peut générer de nouveaux systèmes CRISPR, des systèmes d'édition génétique composé d'un ARN guide et d'une protéine Cas, utilisés pour cibler et modifier spécifiquement des séquences d'ADN dans le génome. Il ouvre ainsi la voie à la création de diversité biologique à partir de modèles génératifs.
- Conception générative : Evo peut non seulement générer des séquences à l'échelle moléculaire, mais également à l'échelle du génome entier, offrant des capacités de conception sans précédent.
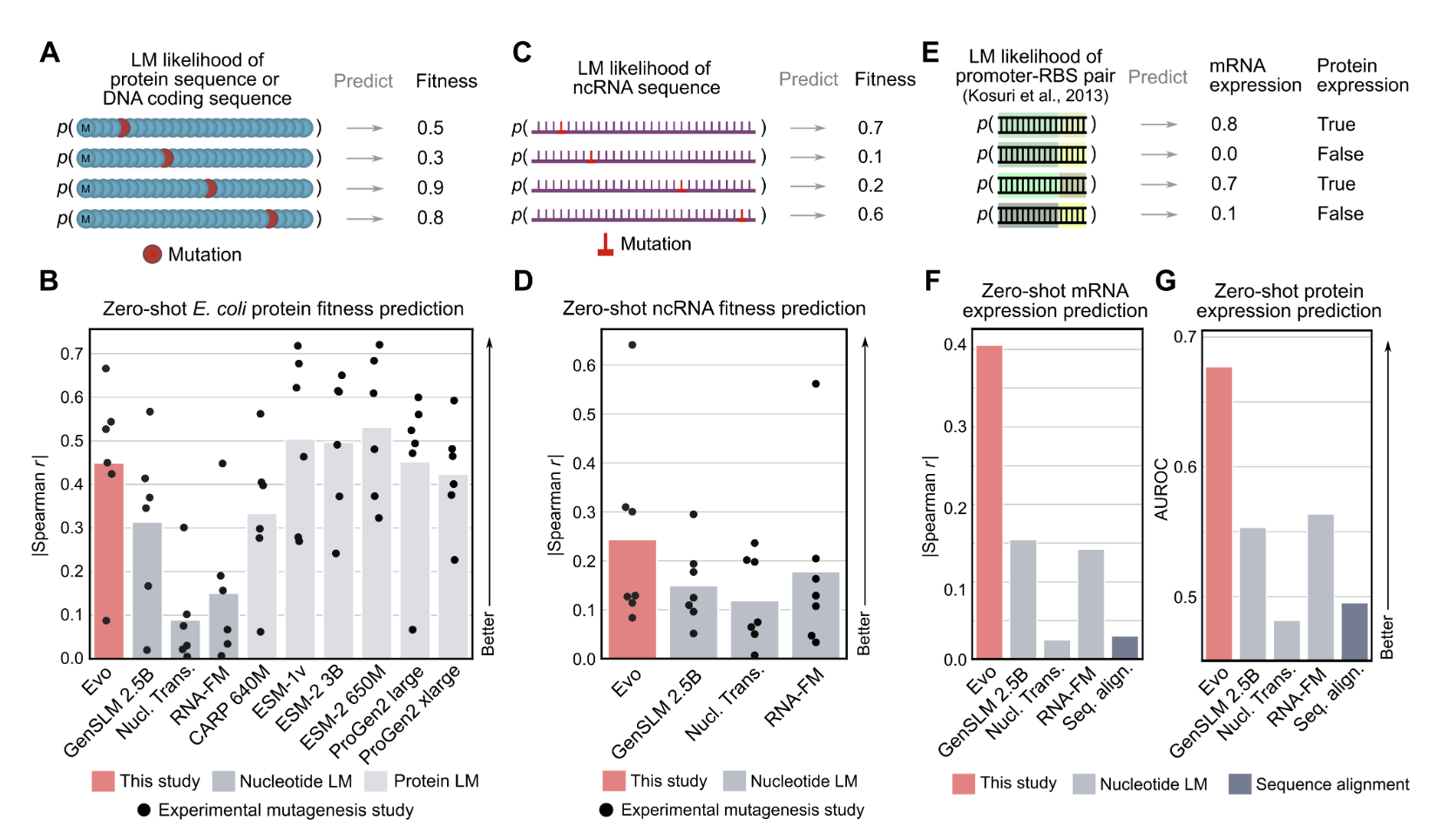
Evo effectue une prédiction de fonction zéro-shot pour les protéines, les ARN non codants et l'ADN régulateur
Perspectives
Evo est le premier du genre à prédire et générer des séquences d'ADN à l'échelle du génome entier avec une résolution mononucléotidique, marquant un tournant dans la modélisation de séquences biologiques. Selon les chercheurs, il a le potentiel d’accélérer la découverte et la compréhension dans les sciences (biologie, chimie, science des matériaux...), ainsi que d’être appliqué à des problèmes concrets comme la découverte de médicaments, l’agriculture et la durabilité.Ils soulignent l'importance de garantir un déploiement sécurisé de modèles ADN à grande échelle comme Evo, et ce, dans l'intérêt de l'humanité. Dans leur article, ils proposent une discussion approfondie sur les risques potentiels et les mesures de précaution à prendre.
Le modèle est disponible sur HuggingFace, GitHub, via l'API Together et<Playground.
Références : Blog Together AI
Article : "Sequence modeling and design from molecular to genome scale with Evo"en prépublication sur BiorXiv, https://doi.org/10.1101/2024.02.27.582234
Auteurs et affiliations :
- Eric Nguyen ( Stanford University, Arc Institute)
- Michael Poli ( TogetherAI)
- Matthew G. Durrant ( Stanford University)
- Armin W. Thomas ( Stanford University)
- Brian Kang (Stanford University)
- Jeremy Sullivan (Arc Institute)
- Madelena Y. Ng (Stanford University)
- Ashley Lewis Stanford University)
- Aman Patel ( Stanford University)
- Aaron Lou (Stanford University)
- Stefano Ermon (Stanford University, CZ Biohub)
- Stephen A. Baccus ( Stanford University)
- Tina Hernandez-Boussard (Stanford University)
- Christopher Ré ( Stanford University)
- Patrick D. Hsu ( Stanford University, University of California, Berkeley)
- Brian L. Hie (Stanford University, Arc Institute)