Dans le cadre d'une recherche collaborative entre le centre de recherche allemand Helmholtz Zentrum München (HZM) et Facebook AI, un modèle d'intelligence artificielle a été conçu afin de prévoir les effets des combinaisons de médicaments, leurs dosages, leur planification. Ce modèle pourra être également utile dans plusieurs types d'intervention comme l'invalidation ou la suppression d'un gène. Ce modèle baptisé Compositional Perturbation Autoencoder (CPA) est en licence open-source et comprend une API ainsi qu'un pack Python.
Une recherche pour parfaire les combinaisons de médicaments
Afin de soigner les tumeurs malignes de patients atteints de cancer, une combinaison de médicaments est préparée en amont. Le CPA est la solution proposée par Facebook AI et le Helmholtz Zentrum München pour trouver une combinaison efficace de médicaments existants avec un dosage approprié. Une problématique de taille lorsque l'on sait que les possibilités peuvent osciller entre 5 000 et 19 milliards de combinaisons.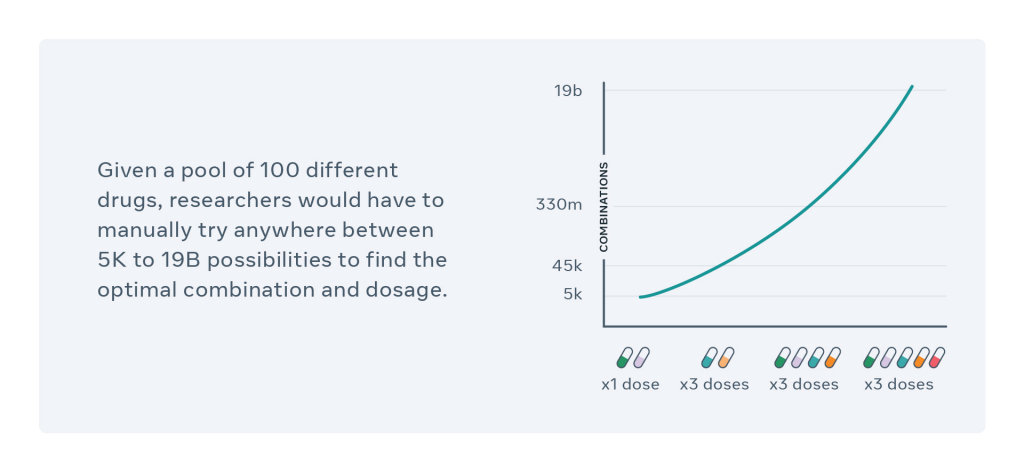 Cette problématique a été le point de départ d'un long travail dont l'ensemble des détails est proposé dans une publication parue le 14 avril dernier. Ces travaux sont le prolongement logique de ceux déjà réalisés autour de méthodes computationnelles liées aux interactions médicamenteuses incluses dans l'ensemble de données d'apprentissage.
Cette problématique a été le point de départ d'un long travail dont l'ensemble des détails est proposé dans une publication parue le 14 avril dernier. Ces travaux sont le prolongement logique de ceux déjà réalisés autour de méthodes computationnelles liées aux interactions médicamenteuses incluses dans l'ensemble de données d'apprentissage.
Ces recherches ont notamment été menées par Mohammad Lotfollahi, Anna Klimovskaia Susmelj, Carlo De Donno, Yuge Ji, Ignacio L. Ibarra, F. Alexander Wolf, Nafissa Yakubova, Fabian J. Theis et David Lopez-Paz, tous auteurs de la publication citée précédemment.
La solution : l'apprentissage auto-supervisé
L'apprentissage auto-supervisé est la solution sur laquelle se sont penchés Facebook AI et le Helmholtz Zentrum München afin de réduire le champ des possibles dans le choix des potentielles combinaisons de médicaments correctement dosées.Un problème qu'ont soulevé plusieurs chercheurs et scientifiques concerne le séquençage de l'ARN en cellule unique. Ce type de séquençage est utilisé comme méthode de mesure des expressions géniques ARN des cellules individuelles au niveau moléculaire. Grâce à cela, il est possible d'étudier les effets des différentes perturbations comme les combinaisons de médicaments ou la suppression d'un gène.
Jusqu’ici, il n’existait pas d’approche efficace permettant de prédire les effets des combinaisons de médicaments inédites et d’autres perturbations. Avec les séquençage d'ARN en cellule unique, des chercheurs ont développé et publié des ensembles de données utilisant cette technique afin que les recherches biomédicales puissent progresser.
Mais tout l'intérêt de ces données, c'est qu'elles soient analysées afin d'en tirer le plus de bénéfices et limiter les combinaisons possibles. Les modèles de machine learning peuvent être une solution pour réaliser ce genre d'analyse mais il faut qu'ils puissent généraliser et extrapoler les aspects intéressants d'une structure de données sans s'appuyer sur des données d'apprentissage caractérisées en vue de réaliser des prévisions portant sur de nouvelles conditions.
La solution conçue par Facebook AI et le Helmholtz Zentrum München consiste à utiliser l'auto-encodage. L'ordinateur synthétise les données pour établir des modèles de prévision utiles. Cette technique d'apprentissage auto-supervisée vise à compresser et décompresser les données, le tout, basé sur les vecteurs d'expression génique non caractérisés de différentes pathologies
Le système repose sur l'isolement des principales caractéristiques d'une cellule comme les effets d'un médicament sur celle-ci, d'un dosage particulier, d'une planification, d'une combinaison, d'une suppression de gène ou d'un type de cellule spécifique. Le modèle d'auto-encodage recombine de manière autonome ces caractéristiques pour prévoir leurs effets sur les expressions géniques de la cellule.
Le modèle est entraîné de tel sorte que le résultat du décodeur corresponde à l’expression génique initialement disponible dans l’ensemble des données de séquençage de l’ARN en cellule unique. On retrouve alors trois étapes caractéristiques à l'apprentissage de ce modèle :
- Le réseau d'encodeur transforme l'expression génique associée à une cellule dans l'ensemble de données en une "représentation de cellule". Un réseau d’intégration traduit le traitement appliqué à cette cellule en une "représentation de traitement".
- En combinant ces représentations de cellule et de traitement, une représentation du goulot d'étranglement est réalisée.
- Un réseau décodeur traduit la représentation du goulot d’étranglement en un vecteur d’expression génique.
Le CPA : une technique d'auto-supervision
Le CPA utilise une technique d'auto-supervision afin d'observer les cellules traitées (celles composant les tumeurs par exemple) avec une quantité limitée de combinaisons de médicaments et prévoit les effets des combinaisons inédites. Imaginons que nous avons quatre types de cellules que nous allons nommer A, B, C et A+B et que nous possédons des données qui permettent de savoir comment les médicaments réagissent sur chacune de ces cellules. Grâce au CPA, les docteurs peuvent déduire l'impact que chaque médicament a sur chaque type de cellule et va même recombiner ces données afin d'extrapoler les combinaisons A+C et B+C ou même les interactions entre A+B et C+D. Le CPA peut générer des hypothèses, orienter les processus de conception expérimentale de nouveaux médicaments mais aussi cibler les combinaisons intéressantes afin de réduire les millions voire les milliards de combinaisons possibles, ce qui évite aux chercheurs de tester l'ensemble de ces combinaisons. Le CPA représente alors un gain de temps non négligeable en plus de proposer une expertise concrète.
Le CPA peut générer des hypothèses, orienter les processus de conception expérimentale de nouveaux médicaments mais aussi cibler les combinaisons intéressantes afin de réduire les millions voire les milliards de combinaisons possibles, ce qui évite aux chercheurs de tester l'ensemble de ces combinaisons. Le CPA représente alors un gain de temps non négligeable en plus de proposer une expertise concrète.
Ce modèle d'auto-encodage a été appliqué à cinq ensembles de données de séquençage de l'ARN contentant des mesures et des résultats pour différents médicaments, dosages et autres perturbations observées sur les cellules cancéreuses. Ses performances ont été mesurées à l'aide d'un indicateur R2 représentant la qualité des prévisions concernant l'expression des gènes individuels.
Les résultats sont les suivants : les scores R2 sont restés constants en ce qui concerne les ensembles d'apprentissage et de test et ont augmenté pour les ensembles hors distribution. Cela signifie que les prévisions du CPA concernant les effets des combinaisons clés de médicaments et des dosages sur les cellules cancéreuses correspondent de manière fiable à celles obtenues dans l’ensemble de données de tests.
Pour tester les limites du modèle, la quantité de données d’apprentissage a été réduite et la quantité de données hors distribution a été augmentée. Malgré une diminution considérable des performances, les prévisions étaient loin d’être aléatoires.


